Learn to Fly
Was andere sich auf das Brot schmieren, steht in Robert Zinzens Labor Eimerweise: Grafschafter Rübensirup. Dazu dutzende Liter Apfelsaft und jede Menge Tüten Haushaltszucker. Der Mann hat einen gesegneten Appetit, würde manch einer denken. Fehlanzeige – aus diesen Zutaten entsteht eine ganz besondere Nahrung. Ein feinmaschiges Netz vor der Tür eines kleinen Extraraums verhindert, dass Robert Zinzens Forschungsobjekte (sie sind genetisch modifiziert) ausbüxen. An der Wand hängen haushaltsübliche Fliegenfänger – für die, denen das bereits gelungen ist.
In der Taufliege, Drosophila, untersucht der Forscher, wie Gene bei der embryonalen Entwicklung des Nervensystems reguliert werden. „Trotz identischer Erbsubstanz entsteht aus einer einzigen befruchteten Zelle ein komplexer Organismus mit wahnsinnig unterschiedlichen Zellen und Geweben, die hochspezialisierte Aufgaben übernehmen. Woher wissen Zellen in der embryonalen Entwicklung, was sie mal werden sollen?“, bringt der Entwicklungsbiologe die für ihn zentrale Frage auf den Punkt. Seit März 2013 leitet er am Berlin Institute for Medical Systems Biology (BIMSB) des MDC die Nachwuchsgruppe „Systembiologie der Differenzierung von neuronalen Zellen und Geweben“.
Leberzelle oder Nervenzelle: Darüber entscheiden die Transkriptionsfaktoren
Ob eine Zelle sich beispielsweise zu einer Leber- oder Nervenzelle entwickelt, regelt unter anderem eine Gruppe von Proteinen – die Transkriptionsfaktoren (TFs). Sie docken an bestimmten DNA-Abschnitten im Erbgut an und regulieren damit unterschiedliche Ableseprogramme, die Transkription. Im Zusammenspiel mit anderen TFs steuern sie, welche Informationen abgelesen werden, und da verschiedene Zellen verschiedene TFs beherbergen, besitzen sie auch unterschiedliche Transkriptionsprogramme. So können genomweit hunderte von TFs kombinatorisch zehntausende von Genen an- oder abschalten und die Entwicklung und Existenz des Organismus garantieren. Für die Feinabstimmungen, beispielsweise wann, wo, und wie stark eine Information abgelesen wird, ist in der Regel das genaue Zusammenspiel zwischen den TFs und den Andockstellen – das sind die Cis-regulatory Elements (CRMs) – verantwortlich. Kleine Unterschiede in den verfügbaren TFs können so große Auswirkungen auf die Transkription bestimmter Gene haben.
Sobald die Transkription im Taufliegenembryo angekurbelt wird, beginnt auch schon die Spezifizierung und Differenzierung der Zellen, und binnen weniger Stunden bilden sich aus einem Pool pluripotenter Zellen differenzierte Komponenten des Nervensystems. Die kurze Entwicklungsdauer, genauso wie der gute genetische und biochemische Zugang, sind Gründe, warum Robert die Insekten für ein ideales Forschungsmodell hält. „Wir untersuchen bestimmte molekulare Abläufe, die entwicklungsbiologisch von der Fliege zum Menschen konserviert sind und darum Rückschlüsse zulassen“, erklärt der Wissenschaftler.
Welche Gene von welchen Transkriptionsfaktoren durch welche Cis-regulatory Elements im Einzelnen getrieben werden bei der Entstehung des Nervensystems, darum geht es dem Biologen nur vordergründig. Die eigentliche Herausforderung liegt darin, genomweit die Netzwerke und das Zusammenspiel der TFs und CRMs zu entschlüsseln:
„Entwicklungsbiologen konnten bislang oftmals nur herausfinden, wie einzelne Gene reguliert werden – nun sind wir endlich technologisch in der Lage zu fragen, wie das Genom reguliert wird und wie dies die Entwicklung steuert.“ Dazu verfolgt er einen integrativen Ansatz: gestützt auf das experimentell bestens erforschte Drosophila-Modell nutzt er den mathematisch-analytischen Charakter der Systembiologie, um Hypothesen aufzustellen und experimentell zu verifizieren. Eines der Ziele ist, einen Atlas der Regulationsstruktur der Nervensystementwicklung zu erstellen. Besonders wichtig ist, wie sich diese Regulationsstrukturen während der Entwicklung verändern und anpassen und wie sich verschiedene Zelltypen daraufhin unterscheiden. „Was wir aber wirklich auf lange Sicht wollen, ist ein mathematisches Modell der Gewebeentwicklung, das nicht nur erklärt, sondern es uns ermöglicht vorauszusagen, wie bestimmte Störungen und Änderungen diese Entwicklung beeinflussen, und wie sich zum Beispiel synthetische Genkonstrukte verhalten würden“, betont der Forscher.
Robert Zinzen ist außerdem dabei, eine Fly-Facility (Fliegen-Plattform) am MDC aufzubauen. So können auch Arbeitsgruppen vom Know-how und der technischen Ausstattung seines Labors profitieren, die keine Erfahrung mit Drosophila haben. Auch wenn der Modellorganismus für die Forschung viele Vorteile hat und zudem recht günstig ist, so ist die Zucht und Haltung der Insekten teils etwas arbeitsintensiv. Embryonen können nicht eingefroren werden. Fliegenstämme müssen in kleinen Röhrchen gehalten und jeden Monat umgesetzt werden. In einer speziellen Fliegenküche wird die Nahrung extra eingekocht. „Was Drosophila für mich aber trotzdem so attraktiv macht – und ich denke auch für andere Arbeitsgruppen – ist die experimentelle Vielfalt, Eleganz und Simplizität mit der man Fruchtfliegen manipulieren kann, und all das in einem sich entwickelnden, relativ komplexen Organismus.
Die Drosophilazucht hat Tradition auf dem Campus Buch
Mit seiner Drosophilazucht knüpft Robert an die Arbeit eines berühmten Wissenschaftlers auf dem Campus Buch an. Am Kaiser-Wilhelm-Institut für Hirnforschung (KWI) nutzte Radiobiologe und Genetiker Nikolai Timoféeff-Ressovsky bereits vor fast 90 Jahren, Drosophila als Untersuchungsmodell für seine Forschung. Mit Hilfe von Röntgenstrahlen erzeugte er Mutationen und untersuchte die genetischen Veränderungen. 1935 veröffentlichte er mit seinen Kollegen Karl Günter Zimmer und Max Delbrück das berühmte „Grüne Buch“, auch „Grünes Pamphlet“ genannt, mit dem Titel „Über die Natur der Genmutation und der Genstruktur“.
Auf dem traditionsreichen Campus weiß sich Robert Zinzen in guter Gesellschaft. „Das MDC ist Weltklasse und bietet sehr gute Bedingungen für Wissenschaftlerinnen und Wissenschaftler. In Deutschland gibt es wenig Vergleichbares, vor allem wenn es um systembiologische Ansätze geht“, betont der 34-jährige. Der gebürtige Amerikaner forschte erst in den USA und wechselte vergangenes Jahr aus der Arbeitsgruppe von Eileen Furlong am EMBL in Heidelberg an das MDC. Die interdisziplinäre Arbeit auf dem Campus ist für ihn ein großes Plus. „Gute Wissenschaft kann selten in Isolation stattfinden, sondern ist auf Kommunikation angewiesen. Vor allem für unsere Fragestellungen ist die Zusammenarbeit mit Mathematikern, Bioinformatikern und anderen Fachrichtungen extrem fruchtbar – daher war das BIMSB am MDC wirklich reizvoll“.
Projekte
Mit seinem Team bearbeitet Robert Zinzen eine Reihe von Projekten. Unter anderem: die Etablierung eines biochemischen Systems zur Isolierung ganz bestimmter Erbgutabschnitte aus sich entwickelnden Embryonen. Mittels transgener Fliegen sollen so CRMs samt gebundener TFs herausgelöst werden – „Ganz wichtig hierbei ist, dass wir solche Untersuchungen immer gewebespezifisch machen. Da sich Gewebe nicht in Isolation, sondern innerhalb des embryonalen Kontexts entwickeln, ist es notwendig die Entwicklungsmechanismen nicht in Kultur, sondern im Embryo zu erforschen“, sagt er.
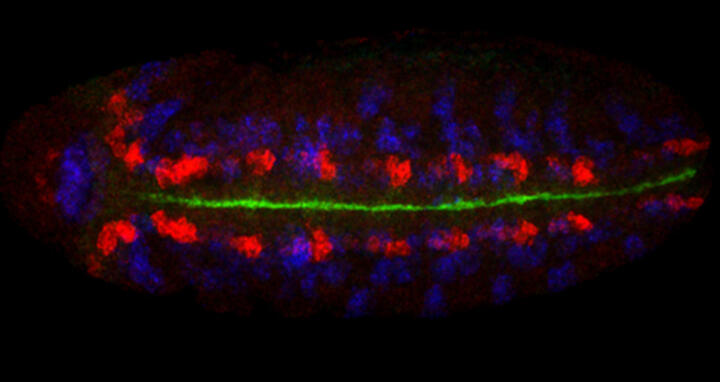
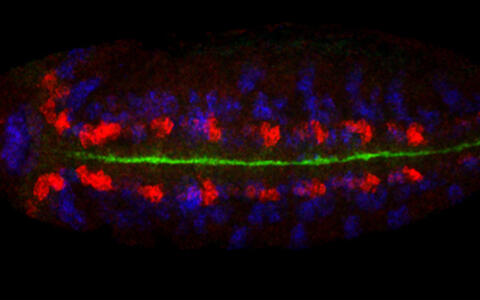
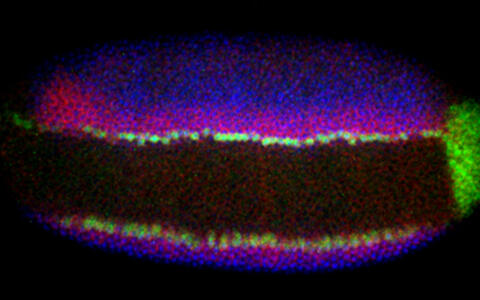
Ein anderes Projekt beschäftigt sich mit der Herstellung spezifischer Antikörper für bestimmte Transkriptionsfaktoren: msh, ind, vnd, sim kontrollieren die Entwicklung verschiedener embryonaler Vorläufer des Nervensystems. Mittels ChIP-Seq (Chromatin ImmunoPrecipitation gefolgt von Sequenzierung) sollen die genomweiten Bindungsprofile sowie deren zeitliche Bindungsdynamik und Kombinatorik untersucht werden.
In einem weiteren Projekt werden Modifikationen von Histonproteinen untersucht. Histone bilden Proteincomplexe um die sich das Erbgut winden und somit verdichten kann. Lange Zeit hielt man sie hauptsächlich für Verpackungseinheiten. Heute weiß man, dass sie entscheidend an der Genregulation beteiligt sind, insbesondere durch chemische Modifikationen der histone tails (Histon-Schwänze). „Bestimmte chemische Modifikationen dieser histone tails sind hoch-indikativ hinsichtlich des Regulationsstatus genomischer Abschnitte – genomweite Modifikationsdaten können uns eine ganz neue Informationsebene der Genomregulation eröffnen“, betont der Wissenschaftler.
Beitragsbild: Gar nicht so unterschiedlich: Entwicklungsbiologisch haben die Taufliege und der Mensch viele Gemeinsamkeiten. Abgebildet ist ein dreifach mutantes Drosophila-Weibchen mit gebogenen statt geraden Flügeln, Barren-förmigen anstelle von runden Augen und gelber Körperpigmentierung. Foto: Robert Zinzen