Verbesserte Methoden für Änderungen am Erbgut
DNA modifizieren können, so wie Redakteure, die an Texten arbeiten: schon seit vielen Jahren träumen Wissenschaftler davon, einzelne Buchstaben des genetischen Codes auszutauschen, Sequenzen in beliebiger Länge zu ändern und große chromosomale Regionen hinzuzufügen, zu löschen, auszutauschen oder neu anzuordnen. Alle diese Vorgänge haben sich im Laufe der Evolution auf natürlichem Weg vollzogen und treten auch auf, wenn die DNA von zwei Elternteilen kombiniert wird, um ein neues Genom zu erschaffen.
In den letzten Jahren wurden äußerst leistungsfähige neue Methoden entwickelt, um Sequenzen von einem Chromosom zu löschen und neue Sequenzen einzufügen. Allerdings sind sie nicht effizient genug, um einzelne Buchstaben des Gencodes problemlos umzuschreiben.
Nun haben die Gruppen von Klaus Rajewsky und Ralf Kühn am MDC eine Methode der Gentechnologie verbessert, die unter der Bezeichnung CRISPR/Cas bekannt ist. Dieses System wurde bereits dazu verwendet, die Veränderung von Zellen aller Arten zu erforschen, darunter auch von befruchteten Eizellen. Durch die Verbesserungen ist jetzt eine Methode entstanden, mit der präzise Mutationen mit hoher Frequenz und Selektivität zum Erbgut hinzugefügt werden können. Die Arbeit, die am 24. März von Nature Biotechnology online veröffentlicht wurde, wird voraussichtlich tiefgreifende Auswirkungen auf die Erforschung von Erkrankungen sowie möglicherweise auch auf die Entwicklung neuer Therapieformen haben.
Hintergrund
Wie viele andere Methoden der Gentechnik basiert auch CRISPR/Cas auf Molekülen, die ursprünglich in Bakterien gefunden wurden. Diese wurden von den Wissenschaftlern für den Einsatz an Mäusen und anderen Versuchstieren angepasst. Bakterien entwickelten dieses System ursprünglich, um sich selbst gegen Viren zu schützen, die während einer Infektion oft kurze Sequenzen ihrer eigenen DNA in die bakteriellen Genome einschleusen.
Durch eine solche erfolgreiche Einschleusung entstehen zwischen den proteinkodierenden Genen in der Regel Sequenzen, die als Spacer-Regionen bezeichnet werden. Sie bestehen häufig aus kleinen, wiederholten Sequenzen, von denen die Bezeichnung CRISPR abgeleitet wird (für clustered regularly interspaced short palindromic repeats). Bakterielle Moleküle, die als CRISPR-assoziierte Gene oder Cas bezeichnet werden, erkennen CRISPR-Sequenzen und führen einen Bruch des DNA-Doppelstrangs herbei. Cas-Proteine werden durch kurze RNA-Stränge (short guiding (sg) RNA) mit spezifischen Sequenzen zu den Zielorten geführt.
DNA-Doppelstrangbrüche entstehen auf natürlichem Wege und werden außerdem durch Strahlenexposition oder Chemikalien, die die DNA schädigen, verursacht. Wenn solche Brüche im Erbgut eines Organismus vorkommen, müssen sie repariert werden, andernfalls stirbt die betroffene Zelle ab. Dieses Phänomen ist die Basis der Strahlen- und Chemotherapien gegen Krebserkrankungen. Zellen, die sich rasch vermehren (wie die Zellen eines Tumors), haben oft nicht genügend Zeit, die notwendigen Reparaturen vorzunehmen, bevor sie absterben.
Es gibt zwei grundlegende Arten der Zellreparatur: homologe und nichthomologe Reparaturen. Der erste Reparaturtyp ist die homologe Rekombination, die jedoch bei Säugetierzellen deutlich seltener ist. Für die Reparatur wird eine exakte Übereinstimmung zwischen den Sequenzen an den Enden eines „gebrochenen“ Fragments und einer vorhandenen, homologen (oder „übereinstimmenden“) Sequenz in einem Genom benötigt. Wird eine solche Übereinstimmung gefunden, so können Enzyme, die die Rekombination der DNA-Moleküle herbeiführen, ein neues Gensegment zwischen den beiden homologen Regionen eines künstlichen DNA-Moleküls einfügen, das als Reparaturmatrize dient.
Mithilfe der homologen Reparatur können Wissenschaftler präzise Genom-Modifizierungen planen und durchführen. Allerdings laufen diese bei Säugetierzellen nur mit niedriger Frequenz ab. Der zweite Reparaturmechanismus, der als nichthomologes Endjoining (NHEJ) bezeichnet wird, ist bei Säugetierzellen wesentlich effizienter. Er ist jedoch auch weniger präzise, da die freien DNA-Stränge ohne Reparaturmatrize einfach wieder miteinander verbunden werden. Häufig gehen im Laufe des Vorgangs DNA-Sequenzen verloren oder werden zusätzlich eingefügt. Die NHEJ-Reparatur kann also eingesetzt werden, um kurze DNA-Sequenzen zu löschen, ist jedoch nicht geeignet für Modifikationen wie die Insertion oder das Umschreiben von speziellen Gensegmenten. Die Prävalenz der NHEJ-Reparatur war eine zentrale Einschränkung für die Arten von Änderungen, die Gentechniker an Genomen vornehmen konnten.
Das aktuelle Projekt
Mäuse und andere Vielzeller haben keine Cas-Proteine, die einen Bruch im DNA-Doppelstrang verursachen. In den vergangenen Jahren ist es Wissenschaftlern jedoch gelungen, Cas-Proteine und sgRNAs künstlich einzuschleusen, um in bestimmte Gene einzugreifen. So erfährt die Zelle, wo im Genom sie die Cas ausliefern und den Bruch herbeiführen soll.
Kombinationen aus sgRNA und Cas können also einen Bruch in einer Sequenz herbeiführen und umfassende Eingriffe in ein Gen verursachen. Das nichthomologe Reparatursystem unterliegt jedoch Einschränkungen, die sich auf die Art sowie auf die Genauigkeit der möglichen Modifikationen beziehen. Klaus Rajewsky, Ralf Kühn, ihre Kollegen am MDC und andere Gruppen stießen kürzlich auf eine Idee, die diese Situation verändern könnte. Die aktuelle Arbeit wurde zeitgleich mit einer Studie von Klaus Rajewskys ehemaligem Kollegen Hidde Ploegh am Whitehead Institute und MIT in Massachussetts (USA) veröffentlicht, in der ein vergleichbarer Ansatz für ein ähnliches Ergebnis angewendet wird. Da beide Gruppen von der Arbeit der jeweils anderen erfahren hatten, beschlossen die Teams von Klaus, Ralf und Hidde Ploegh die Veröffentlichung ihrer Ergebnisse, die sich gegenseitig bestätigten, miteinander zu koordinieren.
„Zellen müssen Doppelstrangbrüche in ihrer DNA reparieren, um zu überleben“, sagt Klaus Rajewsky. „Wenn sie keine nichthomologe Reparatur durchführen können, müssen sie entweder auf den homologen Reparaturtyp ausweichen, oder sie sterben ab. Wenn es uns also gelingen würde, den nichthomologen Reparaturmechanismus irgendwie zu blockieren, so könnten wir sicherstellen, dass die sgRNA-Abschnitte die Cas genau an einer bestimmten Position einfügen und so genau die Art von Änderung durchführen würden, die wir uns wünschen.”
Die entsprechende Methode gründet darauf, dass die homologe und die nichthomologe Reparatur auf unterschiedlichen Enzymen basieren. Ein Protein mit der Bezeichnung DNA Ligase IV ist entscheidend für die nichthomologe Reparatur und ist ausschließlich in diese involviert. Durch die Blockade dieses Proteins müssten die Wissenschaftler in der Lage sein, Zellen dazu zu zwingen, den anderen Weg einzuschlagen.
Dafür standen verschiedene Methoden zur Verfügung: Das Team könnte einen „Knock-down“ der Ligase IV durchführen, die Enzymaktivität mithilfe von SCR7, eines niedermolekularen Inhibitors hemmen, oder Proteine exprimieren, die die Zersetzung der Ligase IV verursachen. Die erste und zweite Methode führten zu einer vier- bis fünffachen Steigerung der Anzahl der homologen Reparaturvorgänge in den Zellen. Die dritte Methode erwies sich sogar als noch effektiver. Indem Zellen dazu gebrachten wurden, zwei Proteine, E1B55K und E4orf6, zu exprimieren, die zur Zerstörung der Ligase IV führten, erreichten die Wissenschaftler eine achtfache Steigerung der homologen Reparatur. Interessanterweise werden diese beiden Moleküle aus einem Erreger abgeleitet, der als Adenovirus bezeichnet wird. Das Virus entwickelte diese Moleküle als Mittel, um die zellulären Abwehrmechanismen zu überwinden. Das Adenovirus schleust E1B55K und E4orf6 Moleküle in die Zellen ein und diese zersetzen die Ligase IV. Indem diese Moleküle zu dem Vektor hinzugefügt wurden, der eingesetzt wurde, um Cas in Zellen einzuschleusen, gelang es den Wissenschaftlern, ein Multifunktionsinstrument zu entwickeln, das die homologe Reparatur sicherstellen sollte.
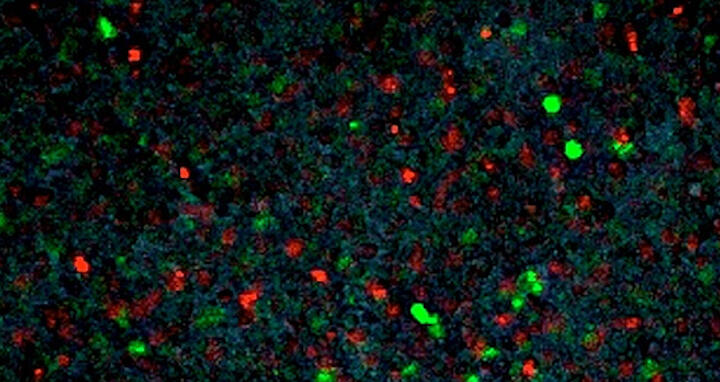
Van Trung Chu, ein promovierter wissenschaftlicher Mitarbeiter in Klaus Rajewskys Gruppe, startete eine Versuchsreihe, um zu prüfen, ob Vektoren, die mit sgRNA, Cas und Adenovirus-Molekülen ausgestattet sind, eine spezifische Sequenz in einer präzisen Position im Genom von menschlichen Zellen aus Zellkulturen einfügen können. Van Trung Chu führte ein „Ampel“-Meldesystem zur DNA-Reparatur ein, das ein grünes Leuchtsignal abgab, wenn das Konstrukt einer homologen Reparatur unterzogen wurde, und ein rotes Signal, wenn der NHEJ-Weg verwendet wurde.
Das Experiment bestand beide Tests. Das Team konnte nachweisen, dass sie eine bestimmte DNA-Region präzise und selektiv ansprechen und anschließend mittels homologer Reparatur jede gewünschte Modifikation umsetzen konnten. Der Rückgang der roten Signale ließ darauf schließen, dass die NHEJ-Reparatur fast vollständig aufgegeben wurde.
Könnte die Methode auch eingesetzt werden, um neue genetisch modifizierte Organismen wie z. B. Mäuse zu entwickeln? Bisher dauerte die Entwicklung solcher Tiere durch Einführung von Änderungen in den embryonalen Stammzellen ein Jahr, manchmal auch zwei. Klaus Rajewsky wandte sich an Ralf Kühn, mit dem er vor zehn Jahren in seinem Kölner Labor zusammengearbeitet hatte. Nach einem Abstecher in München hat Ralf Kühn kürzlich seine eigene Gruppe am MDC gegründet. Er bringt umfassende Erfahrung in der Neuprogrammierung von Stammzellen und der Einführung von genetischen Veränderungen mit, die genutzt werden kann, um Erkrankungen zu erforschen. Außerdem ist er Leiter der Transgenic Core Facility, die Wissenschaftlern dabei hilft, neue Tiermodelle zu entwickeln. Die Einrichtung ist ein Bestandteil des Berliner Instituts für Gesundheitsforschung, wo sie für Gemeinschaftsprojekte von MDC und Charité eingesetzt wird.
Das MDC hat ebenso wie zahlreiche andere Einrichtungen weltweit mit Cas und sgRNAs erfolgreich Modifikationen in befruchtete Eizellen von Mäusen eingeführt. Für das neue System sind keine embryonalen Stammzellkulturen erforderlich. Deshalb wird viel Zeit gespart, indem diese Schritte auf eine einzige Intervention an einzelligen Embryos reduziert werden. Sowohl bei Zelllinien von Mäusen als auch von Menschen ist NHEJ jedoch der vorherrschende Reparaturmechanismus bei einzelligen Embryos. Dadurch wird die Ausbeute der präzise modifizierten Mutanten begrenzt, die in einem beliebigen Experiment erhalten werden. Dieses Problem kann dadurch gelöst werden, dass man die manipulierten Embryos einem niedermolekularen Inhibitor für Ligase IV aussetzt, wie das Team von Hidde Ploegh nachwies. Derzeit wird in der MDC-Einrichtung getestet, ob durch den Einsatz von Proteinen aus Adenoviren noch mehr präzise Mutanten erzeugt werden können. Dafür wird das dort entwickelte „Ampel-“Reportersystem für Maus-Zelllinien eingesetzt.
Weitere Auswirkungen
Wissenschaftler am MDC und an anderen Instituten setzen das CRISPR/Cas-System bereits ein, um neue Zelllinien und Organismen für die Krankheitserforschung zu entwickeln. Sie können das System jetzt verwenden, um Mutationen und andere genetische Veränderungen, die in Erkrankungen involviert sind, wesentlich einfach zu imitieren, ihre biologischen Auswirkungen zu untersuchen und neue Therapieformen zu testen.
Zeitgleich weist Klaus Rajewsky darauf hin, dass diese Arbeit noch weiter reichende Auswirkungen hat, die sofort die Aufmerksamkeit von Forschern und Medien auf sich gezogen haben. Ein zentrales Thema betrifft das Potenzial der Gentechnologie, Änderungen an menschlichen Keimzellen vorzunehmen. Dieser Gedanke ruft eine Reihe von ethischen und moralischen Bedenken wach, die seit Jahren geäußert werden. Bis heute haben die technischen Grenzen des „Genome Editing“ dafür gesorgt, dass beträchtliche Barrieren für den Einsatz der Methode zur Veränderung von humanen Keimzellen bestehen, beispielsweise bei der Korrektur von erblichen Gendefekten. Die Erfolgsquote war angesichts der Prävalenz der nichthomologen Reparatur bei Säugetierzellen relativ gering. Mit anderen Worten: Veränderungen könnten für Zellen tödlich sein oder neue, unerwünschte Defekte verursachen. Im Zuge der Verbesserungen, die mit Sicherheit im Laufe der Zeit noch effektiver werden, wird die Korrektur von Defekten wahrscheinlich äußerst zuverlässig und effizient. Derartige Veränderungen sind für zahlreiche Familien, die von schwerwiegenden Krankheiten betroffen sind, sicherlich wünschenswert. Gleichzeitig könnte die gleiche Methode auch für andere Veränderungen an menschlichen Embryos genutzt werden.
Mitte März zeigte die Veröffentlichung von mehreren Arbeiten, die sich auf die CRISPR/Cas-Technologie beziehen, dass viele technische Hindernisse wahrscheinlich in Kürze überwunden sein werden. Am 19. März veröffentlichten David Baltimore, Keith Yamamoto und 16 weitere renommierte Wissenschaftler einen dringenden Appell zu diesem Thema in der Zeitschrift Science. Das Schreiben riet von jedweden Interventionen an menschlichen Keimbahnen ab, selbst für als dringlich geltende klinische Einsatzzwecke, während die „Wissenschafts- und Regierungsorganisationen noch über die gesellschaftlichen, umwelttechnischen und ethischen Auswirkungen einer solchen Aktivität diskutieren.“ Es wurde hervorgehoben, dass erweiterte Bildungsprogramme und öffentliche Diskussionsforen für eine transparente Forschung zur Untersuchung der Präzision der Methode erforderlich sind, ebenso wie die Gründung einer globalen Gruppe von Experten aus allen relevanten Bereichen, die die Auswirkungen von Manipulationen am menschlichen Genom sorgfältig prüft. Klaus Rajewsky und seine Kollegen werden bei diesen Diskussionen sicherlich eine aktive Rolle spielen.
In der Zwischenzeit, so Klaus Rajewsky, biete das verbesserte System Naturwissenschaftlern das bislang leistungsstärkste Instrument, um die Biologie von Erbkrankheiten an Zellen und Modellorganismen zu untersuchen. So werden weniger Versuchstiere für die biomedizinische Forschung benötigt, außerdem können mit Sicherheit präzisere Einblicke in zahlreiche Erkrankungen gewonnen werden, die die Menschheit bedrohen.
Referenz:
Van Trung Chu, Timm Weber, Benedikt Wefers, Wolfgang Wurst, Sandrine Sander, Klaus Rajewsky, Ralf Kühn. Increasing the efficiency of homology-directed repair for CRISPR-Cas9-induced precise gene editing in mammalian cells. Nature Biotechnology (2015), doi:10.1038/nbt.3198
Pressemitteilung des MDC
Ethics of embryo editing divides scientists, Nature (18. März 2015)
Beitragsbild: Dieses Bild zeigt Zellen, die mit dem "Ampel"-Reportermodul transfiziert wurden. Wenn die Sequenz unter Verwendung des NHEJ-Pathways in das Genom platziert wird, werden die Sequenzen unterbrochen und die Zelle gibt ein rotes Signal. Wenn homologe Reparatur verwendet wurde, bleibt die Reihenfolge intakt. Die Zellen fügen die Probe in die Zwischenräume ein und geben ein grünes Signal. Das Bild ist typisch für frühere Experimente mit CRISPR / Cas, als beide Signalwege in Zellen aktiv waren. Die neuen Experimente zeigen, dass die Blockierung des NHEJ-Pathways zu homologer Reparatur führt und die meisten Zellen das grüne Signal aussenden. Foto: Lab Ralf Kühn, MDC