Proteinproduktion & Charakterisierung
Dr. Anja Schütz
Profil
Unsere Mission ist es, Technologieentwicklung und Spitzenforschung durch die aktive Zusammenarbeit mit anderen Technologieplattformen und Forschungsgruppen zu ermöglichen. Die Plattform bietet Lösungen für die individuelle Herstellung von Proteinen für verschiedene Nachfolgeanwendungen wie zum Beispiel:
- Interaktionsstudien
- Hochdurchsatz-Screening-Kampagnen
- Werkzeug-Proteine
- Antigen-/Antikörperherstellung
- Strukturbiologische Projekte
- Biochemische & biophysikalische Charakterisierung
Neben der kundenspezifischen Proteinproduktion ist die Plattform ein idealer Partner, um gemeinsame Forschungsprojekte zu Schwerpunkten wie Proteindesign und Proteincharakterisierung durchzuführen. Wir haben ein starkes Interesse an der Untersuchung von Struktur-Funktionsbeziehungen von Proteinen, die mit menschlichen Erkrankungen im Zusammenhang stehen. Durch die Anwendung verschiedener biochemischer und biophysikalischer Methoden zeigen wir die physikochemischen Eigenschaften der analysierten Proteine auf, eine Voraussetzung für das Verständnis ihrer biologischen Funktion.
Wir stehen in engem Kontakt mit anderen Einrichungen, die Proteine herstellen und nehmen an internationalen Benchmarking-Studien teil, die durch das P4EU Netzwerk initiiert wurden.
Die Technologieplattform ist weiterhin ein etablierter Partner für die Ausbildung von Lehrlingen am MDC und rekrutiert zudem Bachelor- und Masterstudenten. Zukünftig wollen wir vor allem interdisziplinäre Promotionsprojekte fördern, indem wir die Möglichkeit der Mitbetreuung von Doktorarbeiten anbieten.
Sie sind an einer Zusammenarbeit mit uns interessiert? Dann freuen wir uns über Ihre Anfrage!
Team
Alumni
Wir danken unseren ehemaligen Teammitgliedern für ihre aktive Unterstützung.
Berger, Ingrid – Technische Assistentin
Donath, Mandy – Bachelor Studentin
Haake, Fabian – Bachelor Student
Lenski, Ulf – Bioinformatiker
Kabuß, Loreen-Claudine H. – MDC Auszubildende
Kuhnke, Alina – MDC Auszubildende
Kurths, Silke – Technische Assistentin
Mustroph, Mandy – MDC Auszubildende
Radusheva, Veselina – Master Studentin
Rossa, Denise – MDC Auszubildende
Simon, Carolin – Master Studentin
Service & Technologien
Wir haben Standardarbeitsanweisungen entwickelt und legen insbesondere Wert auf die Qualitätskontrolle. Unsere Philosophie ist die enge Zusammenarbeit und der aktive Gedankenaustausch mit unseren Kooperationspartnern, da dies Voraussetzung für eine erfolgreiche Zusammenarbeit ist.
- Service
Die folgenden Dienstleistungen werden von der Plattform angeboten:
Beratung Wissenschaftliche & technische Fragen Molekularbiologie
(1) Konstruktdesign
(2) Klonierung
(3) Ortsspezifische Mutagenese
Proteinherstellung (1) Expressions- & Löslichkeitstests im Kleinmaßstab
(2) Expression im Großmaßstab
(3) Proteinreinigung
Proteincharakterisierung (1) Bestimmung und Optimierung der Proteinstabilität
(2) Untersuchungen von Protein-Ligand-Wechselwirkungen (Ligand = Protein, Nukleinsäure, kleines Molekül)
(3) Oligomerisierungsanalysen
(4) Massenspektrometrische Untersuchung intakter Peptide und Proteine mittels LC/MS TOF
(5) Bestimmung der Proteinstruktur (inklusive Kristallisation und Datensammlung)
(6) Proteinfaltungsanalysen
- Methoden und Technologien
Proteinproduktion
Genexpression
Konstrukt-Screening und Expression im Großmaßstab
Bakterien – E. coli (bis zu 48 L)
Säugetierzellen – HEK293, CHO (Suspension, transient)
Proteinreinigung
ÄKTA-FPLC-Chromatographiesysteme
AC-, SEC-, IEX-, HIC-Säulen &-Harze
Rückfaltung
Proteincharakterisierung
©Agilent Technologies, Inc. Reproduced with Permission, Courtesy of Agilent Technologies, Inc.
TOF LC/MS (Agilent)
Analyse der intakten Proteinmasse
©Wyatt Technology Corporation, Santa Barbara, CA, USA
DynaPro Plate Reader III (Wyatt)
Bestimmung und Optimierung der Protein-Stabilität (DLS/SLS)
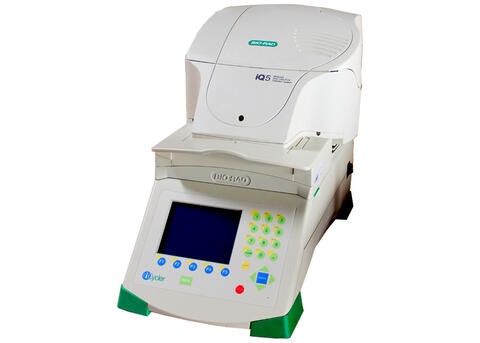
iQ5 RT-PCR (BioRad)
Thermischer Shift-Assay (DSC)
MicroCal PEAQ-ITC (Malvern)
Protein-Liganden-Interaktionsstudien
Chirascan CD Spectrometer (AppliedPhotophysics)
Proteinfaltungsanalyse
Protein Structure Determination
©ARI - Art Robbins Instruments, Crystal Gryphon LCP
Crystal Gryphon LCP
(Art Robbins Instr.)
©FORMULATRIX® - Laboratory Automation Solutions Rock Imager 1000
Rock Imager
4°C und 20°C
(Formulatrix)
Access to synchrotron
(HZB - Bessy)
Falls Sie an anderweitigen proteinbiochemischen Analysen interessiert sind, dann nehmen Sie bitte Kontakt mit uns auf.
Veröffentlichungen
- Begutachtete Publikationen
- Danksagungen
- Oskar Gustafsson, Supriya Krishna, Sophia Borate, Marziyeh Ghaeidamini, Xiuming Liang, Osama Saher, Raul Cuellar, Björn K. Birdsong, Samantha Roudi, H. Yesid Estupiñán, Evren Alici, CI Edvard Smith, Elin K. Esbjörner, Simone Spuler, Olivier Gerrit de Jong, Helena Escobar, Joel Z. Nordin, Samir EL Andaloussi,Advanced Peptide Nanoparticles Enable Robust and Efficient delivery of gene editors across cell types. bioRxiv 2024.11.27.624305; doi: https://doi.org/10.1101/2024.11.27.624305
Roske, Y., Cappel, C., Cremer, N., Hoffmann, P
Roske, Y., Cappel, C., Cremer, N., Hoffmann, P., Koudelka, T., Tholey, A., Heinemann, U., Daumke, O., and Damme, M. Structural analysis of PLD3 reveals insights into the mechanism of lysosomal 5' exonuclease-mediated nucleic acid degradation. Nucleic Acids Res 52, 370-384, 2024. https://doi.org/10.1093/nar/gkad1114
Li X., Wirtz T., Weber T., Lebedin M., Lowenstein E.D., Sommermann T., Zach A., Yasuda T., de la Rosa K., Chu V.T., Schulte J.H., Müller I., Kocks C., Rajewsky K. Precise CRISPR-Cas9 gene repair in autologous memory T cells to treat familial hemophagocytic lymphohistiocytosis. Sci Immunol 9, eadi0042, 2024. https://doi.org/10.1126/sciimmunol.adi0042
Lebedin M., Ratswohl C., Garg A., Schips M., Vazquez Garcia C., Spatt L., Thibeault C., Obermayer B., Weiner J., Moreno Velásquez I., Gerhard C., Stubbemann P., Hanitsch L.G., Pischon T., Witzenrath M., Sander L.E., Kurth F., Meyer-Hermann M., de la Rosa K. Soluble ACE2 correlates with severe COVID-19 and can impair antibody responses. iScience 27, 109330, 2024. https://doi.org/10.1016/j.isci.2024.109330
Zhang J., Sommermann T., Li X., Gieselmann L., de la Rosa K., Stecklum M., Klein F., Kocks C., Rajewsky K. LMP1 and EBNA2 constitute a minimal set of EBV genes for transformation of human B cells. Front Immunol 14: 1331730, 2023. https://doi.org/10.3389/fimmu.2023.1331730
Ratswohl C., Vázquez García C., Ahmad A.U.W., Gonschior H., Lebedin M., Silvis C.E., Spatt L., Gerhard C., Lehmann M., Sander L.E., Kurth F., Olsson S., de la Rosa K. A design strategy to generate a SARS-CoV-2 RBD vaccine that abrogates ACE2 binding and improves neutralizing antibody responses. Eur J Immunol 53(10): e2350408, 2023. https://doi.org/10.1002/eji.202350408
Secker C., Motzny A.Y., Kostova S., Buntru A., Helmecke L., Reus L., Steinfort R., Brusendorf L., Boddrich A., Neuendorf N., Diez L., Schmieder P., Schulz A., Czekelius C., Wanker E.E. The polyphenol EGCG directly targets intracellular amyloid-β aggregates and promotes their lysosomal degradation. J Neurochem 166(2): 294-317, 2023. https://doi.org/10.1111/jnc.15842
M. Broto, M.M. Kaminski, C. Adrianus, N. Kim, R. Greensmith, S. Dissanayake-Perera, A.J. Schubert, X. Tan, H. Kim, A.S. Dighe, J.J. Collins, and M.M. Stevens (2022) Nanozyme-catalysed CRISPR assay for preamplification-free detection of non-coding RNAs. Nature Nanotechnology, 17, 1120-1126.
A.C. McGarvey, W. Kopp, D. Vucicevic, K. Mattonet, R. Kempfer, A. Hirsekorn, I. Bilic, M. Gil, A. Trinks, A.M. Merks, D. Panáková, A. Pombo, A. Akalin, J.P. Junker, D.Y.R. Stainier, D. Garfield, U. Ohler, and S.A. Lacadie (2022) Single-cell-resolved dynamics of chromatin architecture delineate cell and regulatory states in zebrafish embryos. Cell Genomics, 2, 100083.
U. Zinnall, M. Milek, I. Minia, C.H. Vieira-Vieira, S. Müller, G. Mastrobuoni, O.G. Hazapis, S. Del Giudice, D. Schwefel, N. Bley, F. Voigt, J.A. Chao, S. Kempa, S. Hüttelmaier, M. Selbach, and M. Landthaler (2022) HDLBP binds ER-targeted mRNAs by multivalent interactions to promote protein synthesis of transmembrane and secreted proteins. Nature Communications, 13, 2727.
W. Winick-Ng, A. Kukalev, I. Harabula, L. Zea-Redondo, D. Szabó, M. Meijer, L. Serebreni, Y. Zhang, S. Bianco, A.M. Chiariello, I. Irastorza-Azcarate, C.J. Thieme, T.M. Sparks, S. Carvalho, L. Fiorillo, F. Musella, E. Irani, E.T. Triglia, A.A. Kolodziejczyk, A. Abentung, G. Apostolova, E.J. Paul, V. Franke, R. Kempfer, A. Akalin, S.A. Teichmann, G. Dechant, M.A. Ungless, M. Nicodemi, L. Welch, G. Castelo-Branco, and A. Pombo (2021) Cell-type specialization is encoded by specific chromatin topologies. Nature, 599, 684-691.
E. Dias D'Andréa, Y. Roske, G.A.P. de Oliveira, N. Cremer, A. Diehl, P. Schmieder, U. Heinemann, H. Oschkinat, and J. Ricardo Pires (2020) Crystal structure of Q4D6Q6, a conserved kinetoplastid-specific protein from Trypanosoma cruzi. Journal of Structural Biology, 211, 107536.
M. Zech, R. Jech, S. Boesch, M. Škorvánek, S. Weber, M. Wagner, C. Zhao, A. Jochim, J. Necpál, Y. Dincer, K. Vill, F. Distelmaier, M. Stoklosa, M. Krenn, S. Grunwald, T. Bock-Bierbaum, A. Fecíková, P. Havránková, J. Roth, I. Príhodová, M. Adamovicová, O. Ulmanová, K. Bechyne, P. Danhofer, B. Veselý, V. Han, P. Pavelekova, Z. Gdovinová, T. Mantel, T. Meindl, A. Sitzberger, S. Schröder, A. Blaschek, T. Roser, M.V. Bonfert, E. Haberlandt, B. Plecko, B. Leineweber, S. Berweck, T. Herberhold, B. Langguth, J. Švantnerová, M. Minár, G.A. Ramos-Rivera, M.H. Wojcik, S. Pajusalu, K. Õunap, U.A. Schatz, L. Pölsler, I. Milenkovic, F. Laccone, V. Pilshofer, R. Colombo, S. Patzer, A. Iuso, J. Vera, M. Troncoso, F. Fang, H. Prokisch, F. Wilbert, M. Eckenweiler, E. Graf, D.S. Westphal, K.M. Riedhammer, T. Brunet, B. Alhaddad, R. Berutti, T.M. Strom, M. Hecht, M. Baumann, M. Wolf, A. Telegrafi, R.E. Person, F.M. Zamora, L.B. Henderson, D. Weise, T. Musacchio, J. Volkmann, A. Szuto, J. Becker, K. Cremer, T. Sycha, F. Zimprich, V. Kraus, C. Makowski, P. Gonzalez-Alegre, T.M. Bardakjian, L.J. Ozelius, A. Vetro, R. Guerrini, E. Maier, I. Borggraefe, A. Kuster, S.B. Wortmann, A. Hackenberg, R. Steinfeld, B. Assmann, C. Staufner, T. Opladen, E. Ružicka, R.D. Cohn, D. Dyment, W.K. Chung, H. Engels, A. Ceballos-Baumann, R. Ploski, O. Daumke, B. Haslinger, V. Mall, K. Oexle, and J. Winkelmann (2020) Monogenic variants in dystonia: an exome-wide sequencing study. Lancet Neurology, 19, 908-918.
D. Sundaravinayagam, A. Rahjouei, M. Andreani, D. Tupina, S. Balasubramanian, T. Saha, V. Delgado-Benito, V. Coralluzzo, O. Daumke, and M. Di Virgilio (2019) 53BP1 supports immunoglobulin class switch recombination independently of its DNA double-strand break end protection function. Cell Reports, 28, 1389-1399.
H. Behrmann, A. Lürick, A. Kuhlee, H. Kleine Balderhaar, C. Bröcker, D. Kümmel, S. Engelbrecht-Vandré, U. Gohlke, S. Raunser, U. Heinemann, and C. Ungermann (2014) Structural identification of the VPS18 β-propeller reveals a critical role in the HOPS complex stability and function. Journal of Biological Chemistry, 289, 33503-33512.
F. Mayr and U. Heinemann (2013) Mechanisms of Lin28-mediated miRNA and mRNA regulation: a structural and functional perspective. International Journal of Molecular Sciences, 14, 16532-16553.
S. Boivin and D.J. Hart (2011) Interaction of the influenza A virus polymerase PB2 C-terminal region with importin a isoforms provides insights into host adaptation and polymerase assembly. Journal of Biological Chemistry, 286, 10439-10448.
H. Striegl, M.A. Andrade-Navarro, and U. Heinemann (2010) Armadillo motifs involved in vesicular transport. PLoS ONE, 5, e8991.
Nachrichten
Wissenstransfer und Öffentlichkeitsarbeit
Auf dieser Seite finden Sie Informationen zu unseren Transfer- und Öffentlichkeitsarbeitsaktivitäten, wie zum Beispiel eine Strukturgalerie von Proteinen und der Link zu Patenten.
- Protein Datenbank
Hier unten finden Sie unsere Kristallstruktur-Einträge in der Protein Data Bank.
- Patente
Entdecken Sie unsere innovativen Beiträge zum Fachgebiet durch unser Patentportfolio, das über den folgenden Link zugänglich ist.