Kaputtes Werkzeug — kaputtes Herz
Die Analyse der Genome des Menschen hat gezeigt, dass unsere DNA weniger Gene enthält, als allgemein unter Wissenschaftlern angenommen, und dass die meisten eher einem Inselmeer ähneln als kompakten Einzelinseln. Eine Sequenz eines Gens besteht für gewöhnlich aus mehreren proteincodierenden Abschnitten, Exons genannt. Diese wechseln sich mit langen, nichtcodierenden Regionen (Introns) ab. Entfernt man die Introns aus dieser Kette, entsteht eine Boten-Ribonukleinsäure (auch mRNA genannt - vom englischen messanger RNA) mit einem nahtlosen Bereich der Proteincodierung. Dieser Vorgang findet im Spleißosom, einer molekularen Maschine, statt. Oftmals kommt es jedoch hierbei auch zur Entfernung einiger Exons. Zellen bilden dann Varianten eines Proteins mit unterschiedlichen Bausteinen und gewebecharakteristischen Funktionen. Bei diesem Spleißvorgang werden Proteine in eine Art Multifunktionswerkzeug verwandelt. Das kann man sich in etwa so vorstellen: Eine ganze Kiste mit unterschiedlichen Schraubendrehern wird gegen mehrere Bit-Aufsätze ausgetauscht, die alle auf ein Werkzeug passen. Somit lässt sich ein komplexer Organismus mit einer viel kleineren Werkzeugkiste fertigen.
Störungen im Spleißen können dazu führen, dass eine Form für ein Protein und somit ein Werkzeug verloren geht, welches für ein bestimmtes Gewebe benötigt wird. Dies ist bei einer Reihe von schweren Erbkrankheiten der Fall. Seit mehreren Jahren gehen die beiden Forscher Prof. Dr. Norbert Hübner und Prof. Dr. Michael Gotthardt davon aus, dies betreffe auch einige Herzkrankheiten, wie die dilatative Kardiomyopathie (DKM). Bei dieser Herzmuskelerkrankung ist das Herz vergrößert und geschwächt. Ein Drittel aller Todesfälle wegen Pumpversagen des Herzens lässt sich auf DKM zurückführen. Jetzt ist es der Forschungsgruppe um Prof. Dr. Hübner in Zusammenarbeit mit den MDC-Forschern Prof. Dr. Gotthardt, Prof. Dr. Nikolaus Rajewsky, Prof. Dr. Matthias Selbach und Dr. Markus Landthaler gelungen nachzuweisen, dass RBM20-Mutationen eine Herzmuskelschwäche, wie die DKM, wahrscheinlich mit verursachen, indem sie das Spleißen von Molekülen beeinträchtigen, die für das Herz wichtig sind. Die Arbeit der Forschungsgruppe erschien in der Ausgabe des Journal of Clinical Investigation vom 1. August.
Laut Prof. Dr. Hübner haben Störungen beim Spleißen verschiedene Ursachen. „Das Spleißosom muss in der Ziel-RNA die Intron/Exon-Grenzen erkennen und die richtige Sequenz entfernen. Ist nun die Sequenz einer RNA aufgrund von Genmutationen verändert, kann es zu Erkennungsschwierigkeiten kommen. Die Ursache kann jedoch auch in der Spleißmaschinerie selbst liegen. Das Spleißosom besteht je nach Gewebe-/Zelltyp aus unterschiedlichen Bauteilen, die speziell auf die jeweiligen Anforderungen ausgerichtet sind. Defekte bei einem dieser Bauteile können zu gewebespezifischen Problemen beim Spleißen führen“, so Prof. Dr. Hübner weiter.
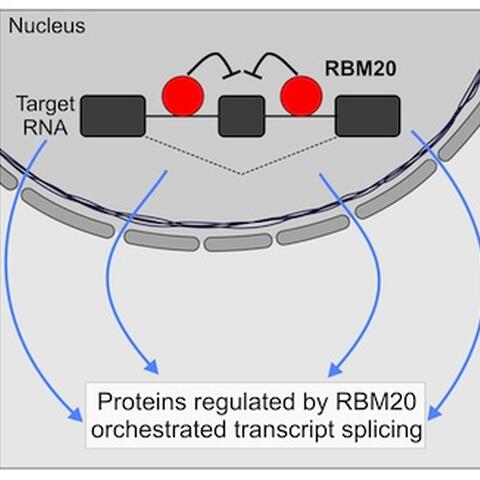
RBM20-Mutationen konnten bereits mit der Entstehung von dilatativen Kardiomyopathien in Verbindung gebracht werden. RBM20 steht für die englische Bezeichnung RNA binding motif protein 20. Das Helferprotein RBM20 bindet an RNA und ist somit mutmaßlich am Spleißvorgang beteiligt. Es wird in großen Mengen im Herzen und in der quergestreiften Muskulatur (Skelettmuskulatur) erzeugt, was darauf hindeutet, dass ihm besondere Funktionen in diesem Gewebe zukommen. 2012 wiesen Prof. Dr. Gotthardt und Prof. Dr. Hübner gemeinsam mit ihren Kollegen die spleißregulierende Funktion von RBM20 bei der Herstellung von Titin, eines sehr großen Proteins, nach. Diesem Protein kommt eine wichtige Funktion im Muskelgewebe zu. Titin fungiert wie eine Art elastische Feder, wenn Muskelzellen kolbenartig kontrahieren und relaxieren. Bei Spleißstörungen entsteht eine Feder, die entweder zu kurz oder zu lang ist und somit die mechanischen Eigenschaften des Muskels verändert. Geschieht das im Herzen, ist es besonders schlimm.
Wissenschaftler wissen jetzt, RBM20 bindet auch an eine Reihe anderer Zielstrukturen, und haben die Aufgaben des Helferproteins RBM20 in unterschiedlichen Vorgängen genau festlegen können. Ausgehend von diesen Arbeiten können Erkenntnisse gezogen werden, wie Proteinmutationen oder Mutationen in den Zielstrukturen, an die sich das Protein bindet, Spleißstörungen bei Molekülen bewirken können, welche die Herzfunktionen stark beeinflussen.
Um diesen Fragen im intrazellulären Bereich nachzugehen, bedarf es Kenntnisse darüber, wie Proteine in einer molekularen Maschine, wie dem Spleißosom, zusammenwirken und wie und wo sie anbinden, z. B. an Ribonukleinsäuren. Die Form der Proteine bietet physikalisch sowie chemisch eine Architektur, dank derer sie mit anderen Molekülen mit passender Struktur interagieren können. Das Helferprotein RBM20 ist höchstwahrscheinlich in der Lage, ein kleines Muster in Ribonukleinsäuren auszulesen, das der molekularen Maschine die Bindungsstelle angibt.
Die Leiterin der aktuellen Studie, Dr. Henrike Maatz, Postdoktorandin in der Forschungsgruppe von Prof. Dr. Norbert Hübner, erklärt: „Wir gingen bisher davon aus, Titin sei die einzige Zielstruktur dieses Hilfsproteins. Ein Sequenzmuster lässt sich jedoch nicht von nur einem Beispiel ableiten. Daher müssen wir untersuchen, ob es auch an andere Ribonukleinsäuren bindet.“
Dafür verwendeten die Wissenschaftler unterschiedliche Ansätze. Mit der so genannten von Dr. Markus Landthaler verwendeten PAR-CLIP-Methode wurde das Protein an RNA-Moleküle „geklebt“. Zur Bestimmung der Protein-RNA-Interaktionen wurde RBM20 aus den Zellen extrahiert und die daran anbindenden Ribonukleinsäuren durch Sequenzierung bestimmt. Nicht unmittelbar an RBM20 bindende Regionen der Ribonukleinsäuren wurden entfernt, sodass sehr kurze Abschnitte verblieben. Hierin sind die Muster zum Anbinden der Ribonukleinsäuren enthalten. Somit war es Dr. Marvin Jens aus der Forschungsgruppe von Prof. Dr. Nikolaus Rajewsky möglich nach einem allgemeinen Muster zu suchen, das von RBM20 erkannt wird. Im Ergebnis zeigte sich dies mit nur vier Nucleotidsequenzen als sehr kurz.
Bei einem anderen Ansatz wurden alle Ribonukleinsäuren, die von Ratten mit gesunden RBM20 produziert wurden, mit denen gegenübergestellt, die von einem Rattenstamm produziert wurde, dessen Tiere dieses Protein fehlte. Die Ergebnisse wurden anschließend mit den Molekülen verglichen, die bei Menschen mit Herzversagen anzutreffen sind.
RBM20-Mutationen könnten sich auf die Fähigkeit des Spleißosom auswirken, Zielstrukturen zu erkennen. Gleichzeitig könnten diese Mutationen die Abläufe der molekularen Maschine selbst beeinträchtigen. Um einen detaillierteren Einblick in die Funktionen des Proteins RBM20 im Spleißosom zu erhalten, wandten sich die Forscher an Dr. Marieluise Kirchner aus dem Labor von Prof. Dr. Matthias Selbach, welches auf die Interaktion zwischen Proteinen spezialisiert ist. Hier erhielten sie wichtige Informationen über die Position des Proteins RBM20 innerhalb der molekularen Maschine, zu den anderen Proteinen im Spleißosom und seinem Beitrag beim Spleißen. Außerdem konnte sie nachweisen, dass Mutationen, wie sie bei einigen Herzpatienten auftraten, die Fähigkeit der Interaktion mit anderen Komponenten der molekularen Maschine beeinträchtigte.
Aus der Synthese dieser Informationen konnten die Wissenschaftler eine Reihe neuer RNA-Ziele benennen. Die Untersuchung des Musters der Zielstruktur, an welche RBM20 anbindet, zeigte, dass diese in den Introns anzutreffen ist. Ihre übliche Funktion an dieser Stelle ist, dem Spleißosom mitzuteilen, das angrenzende Exon zu entfernen und das Molekül zu kürzen. Bei Tieren und Menschen, bei denen dieses Protein fehlerhaft hergestellt wird, wird auch das Exon nicht entfernt. Dadurch entstehen Ribonukleinsäuren mit zusätzlichen Abschnitten, die wiederum eingesetzt werden, um zu lang geratene Varianten der so wichtigen Proteine zu bauen. Bei der Herstellung des Proteins Titin ist somit die Feder nicht ausreichend gespannt, was zu unzureichender Kontraktionsfähigkeit der Muskulatur führt. Dadurch muss das Herz mehr arbeiten und vergrößert sich.
„Durch die Studie konnten viele Erkenntnisse aus vorangegangener Forschung ins Verhältnis gesetzt werden. Zwar war bereits ein Zusammenhang zwischen Mutationen bestimmter anderer Moleküle und der dilatativen Kardiomyopathie hergestellt, wie diese Mutationen jedoch zu diesem Krankheitsbild beitragen, war unbekannt. Jetzt hat sich gezeigt, dass die meisten dieser Moleküle Anbindestellen des Proteins RBM20 sind und wahrscheinlich beim Spleißen Fehler auftreten. Zudem haben wir weitere Zielstrukturen entdeckt, die ebenfalls in gleicher Weise betroffen sein könnten, jedoch mit dem Krankheitsbild noch gar nicht in Verbindung gebracht wurden. Diese Erkenntnisse bieten weitere Ansatzmöglichkeiten, diese komplexe und tödliche Krankheit besser verstehen zu können“, erklärt Prof. Dr. Norbert Hübner.
Beitragsbild: RBM20-Mutationen konnten bereits mit der Entstehung von dilatativen Kardiomyopathien in Verbindung gebracht werden. RBM20 steht für die englische Bezeichnung RNA binding motif protein 20. Foto: AG Hübner, MDC