AG Kühn
Genom-Editierung & Krankheitsmodelle
Profil
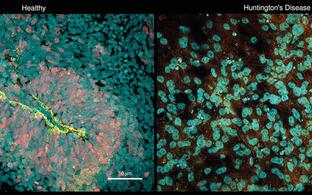
Wir verwenden fortgeschrittene CRISPR-Cas9 Technologien, insbesondere unsere patentierte REPLACE Technik* (Danner et al. 2021), für das Engineering von Genomsequenzen in Säugetierzellen. Mit diesen Werkzeugen generieren wir neue Allele zur Untersuchung als auch der Therapie genetisch basierter Krankheiten in Modellsystemen aus Mensch und Maus.
Im Einzelnen verfolgen wir Forschungsarbeiten zur:
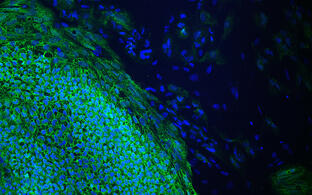
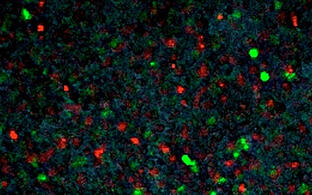
- Generierung neuer Krankheitsmodelle in Mäusen und menschlichen iPS Zelllinien
- Gentherapie in Mausmodellen und Patienten-Zelllinien
- Austausch von Exonabschnitten durch NHEJ Reparatur zur Therapie und Humanisierung von Genen
- Generierung genetischer Mausmodelle in der MDC Serviceeinheit
- Reparatur von Defekten im Genom der C57BL/6 Inzuchtmaus
Genom-Engineering ist eine Schlüsseltechnologie zur Untersuchung von Genfunktionen die zum Verständnis von Krankheitsprozessen beitragen. Die kontinuierliche Innovation von Techniken zur Gen-Editierung bildet eine Grundlage für die zukünftige biomedizinische Forschung.
*Nucleic Acid Sequence Replacement by NHEJ. PCT International Publication Number: WO 2019/122302 A1 (2019).