Wie RNA-bindende Proteine reguliert sind
Wenn Zellen ein bestimmtes Protein brauchen, lesen sie erst einmal das entsprechende Gen ab und schreiben dessen DNA-Sequenz in RNA um (Transkription). „Die Forschung hat sich lange stark auf die dafür nötigen Transkriptionsfaktoren konzentriert. Also darauf, wie die unmittelbare Bildung der RNAs kontrolliert wird. Aber das ist nur Schritt eins der Genexpression. Auch was danach kommt – die posttranskriptionale Regulation – ist ein sehr wichtiger Prozess!“, erklärt Professor Matthias Selbach, Leiter der Arbeitsgruppe „Proteom-Dynamik“ am Berliner Max-Delbrück-Centrum für Molekulare Medizin in der Helmholtz-Gemeinschaft (MDC) und Letztautor einer Studie, die jetzt in der Fachzeitschrift „Molecular Cell“ erschienen ist.
Im nächsten Schritt kommen RNA-bindende Proteine ins Spiel, um die es in der Studie geht. Bei der Translation außerhalb des Zellkerns, im umgebenden Zytoplasma, dient die RNA – jetzt in Form von Boten-RNA – den Ribosomen als Bauplan für den Aufbau von Proteinen aus Aminosäuren. RNA-bindende Proteine sorgen dafür, dass die Boten-RNA aus dem Zellkern transportiert wird, zum Ribosom gelotst, sich stabilisiert und wieder abgebaut werden kann, wenn sie nicht mehr vonnöten ist.
Anders als bei Transkriptionsfaktoren weiß man allerdings erst wenig darüber, wie die rund 1.900 RNA-bindenden Proteine reguliert sind. Meist sind es chemische Modifikationen, die die Funktionen von Proteinen verändern – so können gezielt Phosphatgruppen an einzelnen Aminosäuren angehängt werden (Phosphorylierung). Dadurch ändert sich in der Regel die Aktivität des Proteins.
Wichtig für die Titin-Synthese
In den vergangenen Jahren wurden mehr als 100.000 solcher Phosphorylierungsstellen an Proteinen gefunden. „Doch bei den allermeisten weiß man nicht, was die Phosphatgruppen funktionell bewirken“, sagt Selbach. Zeit also, sich die RNA-bindenden Proteine einmal ganz genau anzusehen. Insbesondere das am MDC intensiv untersuchte RBM20, ein RNA-bindender „Masterregulator“. Er spielt bei der Bildung des variantenreichen, elastischen Proteins Titin im Herzmuskel und damit zum Beispiel bei der Entstehung von Herzmuskel-Erkrankungen eine entscheidende Rolle. Dr. Carlos Henrique Vieira e Vieira, Erstautor der Studie, verwendete dazu die am MDC von Professor Markus Landthaler entwickelte RNA-Interactom-Erfassung – kurz RIC – und erweiterte sie zu einer quantitativen Methode (qRIC).
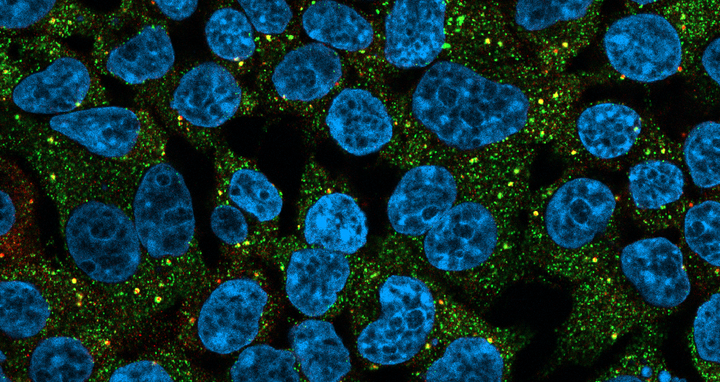
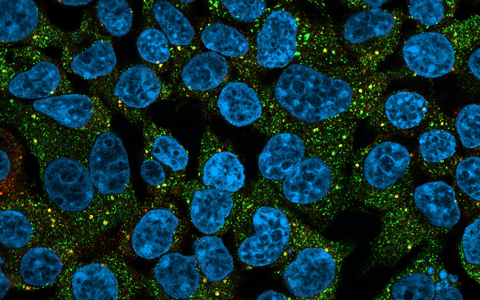
Das Bild zeigt menschliche Zellen mit einer krankheitsauslösenden RBM20-Mutante: Hellblau sind die Zellkerne dargestellt, grün das RBM20-Protein und rot das Protein MOV10. Es markiert granuläre Organellen in den Zellen, die RNA enthalten, das RNA enthaltende, granuläre Organellen in den Zellen markiert.
Normalerweise sind Proteine während ihres Zusammenspiels mit der RNA durch schwache Kräfte wie elektrostatische Wechselwirkungen und Wasserstoffbrückenbindungen verbunden. „Durch UV-Licht frieren wir quasi diesen Zustand der Zelle ein – und erst dann schließen wir sie auf“, erklärt Matthias Selbach. Dazu werden lebende Zellen mit UV-Licht bestrahlt. Alle Proteine in den Zellen, die gerade sehr dicht an RNA-Molekülen sitzen, werden dadurch stabil an diese gebunden. Crosslinking nennen die Forschenden das.
Mehr als hundertmal fündig
Anschließend werden die RNAs samt der an sie gebundenden Proteine herausgefischt, gereinigt und aufgetrennt. Im Massenspektrometer werden die Moleküle ionisiert und zerfallen in charakteristische Bruchstücke. Und darüber können die Forschenden nun auch rekonstruieren, an welchen Stellen der Aminosäurekette der RNA-bindenden Proteine Phosphatgruppen sitzen. Quantitativ wird die Methode, indem man den prozentualen Anteil der phosphorylierten mit den entsprechenden freien Proteinen (ohne Phosphatgruppen) vergleicht.
„Mit dieser quantitativen Methode haben wir über 100 Phosphorylierungsstellen mit regulatorischem Potenzial an verschiedenen RNA-bindenden Proteinen entdeckt. Darunter haben wir auch solche wiedergefunden, von denen bereits bekannt ist, dass sie regulieren“, sagt Carlos Vieira. Dass Phosphatgruppen an diesen Proteinen tatsächlich regulatorische Effekte haben, wiesen die Forschenden in Kooperation mit dem RBM20-Experten Professor Michael Gotthardt nach, der ebenfalls am MDC arbeitet. Sie brachten RBM20-Mutationen in Zellkulturzellen ein, die entweder an bestimmten Stellen nicht phoshoryliert werden konnten oder statt Phosphat andere chemische Gruppen trugen.
Alternatives Spleißen gestört
Wir haben Phosphorylierungsstellen identifiziert, die tatsächlich die Regulation von RBM20 beeinflussen.
Das Resultat: bei RBM20-Mutanten war das alternative Spleißen gestört. Diesen raffinierten Trick der Natur reguliert RBM20. Er erlaubt, dass ein einzelnes Gen den Bauplan für viele Varianten eines Proteins liefert – in diesem Fall des Titins, das für das Herz so wichtig ist. „Die Experimente mit den Mutanten waren für mich ein Wendepunkt. Ich war überrascht, wie eindeutig die Ergebnisse sind. Denn in der Regel kann bei solchen Experimenten viel schief gehen“, betont Viera.
„Wir haben demnach Phosphorylierungsstellen identifiziert, die tatsächlich die Regulation von RBM20 beeinflussen“, erläutert Matthias Selbach. „Es ist der erste Hinweis darauf, dass diese Stellen funktionell wichtig sind. Ob es für den Herzmuskel oder dessen krankhafte Veränderungen wie bei Kardiomyopathien entscheidend ist, können wir aber noch nicht sagen.“
Die qRIC-Methode ist jedenfalls anscheinend die ideale Ergänzung zu routinemäßigen Proteom-Analysen. Denn diese erfassen zwar die Gesamtheit der möglichen Modifikationen von Proteinen, die durch Phosphorylierungen entstehen. Sie können aber keinen Einblick in deren mögliche Funktion geben.
Text: Catarina Pietschmann
Weiterführende Informationen
- Porträt Matthias Selbach: Der Proteinfahnder
- Porträt Michael Gotthardt: Der Perfektionist
- Neuer Schwung für schwache Herzen
Literatur
Carlos Henrique Vieira e Vieira et al. (2022): „Proteome-wide quantitative RNA interactome capture (qRIC) identifies phosphorylation sides with regulatory potential in RBM20“. Molecular Cell, DOI: 10.1016/j.molcel.2022.03.024
- Max-Delbrück-Centrum für Molekulare Medizin (MDC)
-
Das Max-Delbrück-Centrum für Molekulare Medizin in der Helmholtz-Gemeinschaft gehört zu den international führenden biomedizinischen Forschungszentren. Nobelpreisträger Max Delbrück, geboren in Berlin, war ein Begründer der Molekularbiologie. An den MDC-Standorten in Berlin-Buch und Mitte analysieren Forscher*innen aus rund 60 Ländern das System Mensch – die Grundlagen des Lebens von seinen kleinsten Bausteinen bis zu organübergreifenden Mechanismen. Wenn man versteht, was das dynamische Gleichgewicht in der Zelle, einem Organ oder im ganzen Körper steuert oder stört, kann man Krankheiten vorbeugen, sie früh diagnostizieren und mit passgenauen Therapien stoppen. Die Erkenntnisse der Grundlagenforschung sollen rasch Patient*innen zugutekommen. Das MDC fördert daher Ausgründungen und kooperiert in Netzwerken. Besonders eng sind die Partnerschaften mit der Charité – Universitätsmedizin Berlin im gemeinsamen Experimental and Clinical Research Center (ECRC) und dem Berlin Institute of Health (BIH) in der Charité sowie dem Deutschen Zentrum für Herz-Kreislauf-Forschung (DZHK). Am MDC arbeiten 1600 Menschen. Finanziert wird das 1992 gegründete MDC zu 90 Prozent vom Bund und zu 10 Prozent vom Land Berlin.